昇思 MindSpore 开源社区将于 2025 年 12 月 25 日在杭州举办昇思人工智能框架峰会。本次峰会的昇思人工智能框架技术发展与行业实践论坛将讨论到昇思MindSpore 大模型技术进展与实践,并将设有昇思 AI for Science(AI4S)专题论坛。本文对 AI4S 团队开发的 MindSpore Protenix 蛋白质结构预测模型的性能与优化进行了深入解读,揭示了如何实现该模型的训练与推理性能的提升。
背景
蛋白质结构预测是现代生命科学的圣杯之一。虽然AlphaFold2等AI工具已实现单体蛋白结构的高精度预测,但整个领域仍面临两大核心瓶颈:
第一,预测准确性仍存在系统性盲区。当前模型对蛋白质动态构象、翻译后修饰状态、膜蛋白环境以及多链复合物组装等关键场景的预测精度严重不足。模型在MSA信息稀疏时(如人工设计蛋白、孤儿蛋白)性能会断崖式下跌,本质上仍是基于进化关联的“模式外推”而非真正的物理规律学习。
第二,计算复杂性成为应用壁垒。最先进的预测模型需要同时处理数千条同源序列的MSA信息,单次推理就需数十GB显存和数小时GPU时间。对于需要高通量扫描的工业场景或更大尺度的复合物预测,算力需求呈指数级增长。这使得前沿技术难以转化为普惠工具,学术实验室和中小企业常因算力门槛而被排除在创新循环之外。
这两个问题相互缠绕:要提升对复杂场景的预测精度,往往需要更庞大的模型和更丰富的输入特征,而这又会进一步推高计算成本,形成难以突破的技术闭环。
昇思 MindSpore 的 AI for Science 方案详解
昇思 MindSpore 通过软硬件协同优化及高效的 NPU 计算能力,为行业提供了高性能的自主创新 AI 解决方案,大幅加速蛋白质研究进程并降低计算成本。我们实现了蛋白质结构预测模型 Protenix 的 MindSpore 框架版本,并在昇腾硬件平台上实现了高性能的训练和推理。为应对大规模蛋白质结构预测的高计算需求,本项目充分利用 MindSpore 框架的计算图优化能力与昇腾处理器的硬件优势,在完全继承了模型推理精度的同时,又显著提升了模型性能。
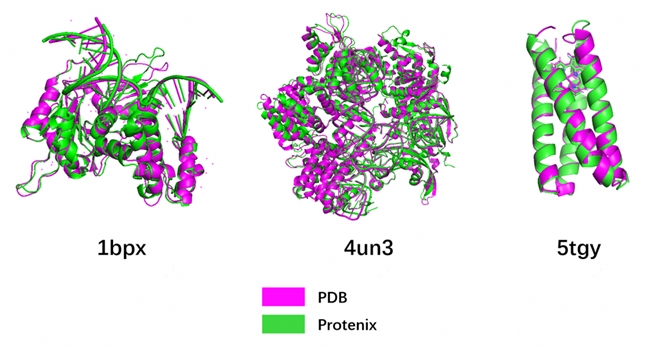
图1 MindSpore Protenix蛋白质结构预测模型的推理效果
在本文所描述的调优策略下,模型在昇腾A2 64G 单卡上可达到 768 的最大训练长度,并且最大单卡推理长度超过 3000;以下是相应的具体训推时间:
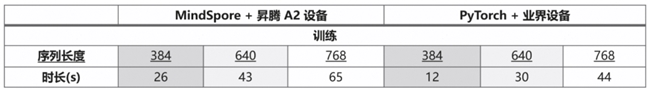
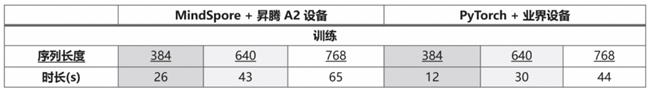
2.1 模型训练优化
重计算(Recompute)优化
在深度模型训练中,显存占用通常可分为静态显存(Static Memory)与动态显存(Dynamic Memory)两个部分。对于 Protenix(AF3 类结构模型) 这类高度依赖几何结构建模的网络而言,其瓶颈并非权重规模,而是激活值数量极大、计算路径复杂、依赖大量三元(i,j,k)结构相关中间张量。通过在前向传播阶段不保存部分激活值,而是在反向传播需要梯度时重新执行对应的前向计算,即可显著降低显存占用。
PyTorch 版本 Protenix 中已经大量使用了重计算来缓解激活膨胀的问题。然而受限于硬件显存容量限制、模型关键结构适配不足,以及考虑到 MindSpore 对动态 shape 的静态优化与 PyTorch 有一定差异后,我们在 MindSpore 版本中对重计算策略做了更细粒度的优化。
如下图红框处所示,a 为未优化前显存占用曲线,可以看到在红框处达到峰值。通过分析可以确定此处位置用于计算 smooth_lddt_loss,因此将这个部分单独进行重计算后就得到了下图的结果,此处峰值由 55G 下降到 20G 以内。
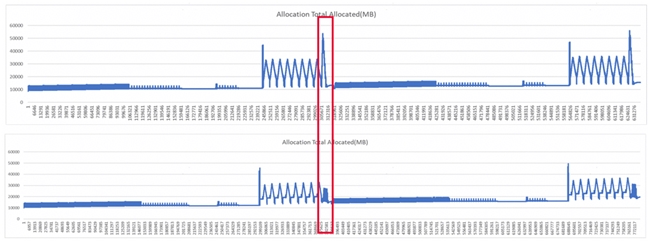
针对性重计算设计
在 MindSpore 实现中,我们分别对核心模块进行了独立的重计算包装,以精确控制激活缓存范围并最大化释放显存。首先是针对 Triangle Attention 的重计算,Triangle Attention 在 AF3 / Protenix 中是最重要的结构依赖模块之一,其 Q/K/V 计算与 pair-wise 三元交互的复杂度为 O(N^3) ,随着序列的增长会产生大量中间激活,在昇腾平台上,由于当前暂时缺乏对等的 fused kernel(如 FlashAttention-like kernel),Triangle Attention 的激活会占用更大量的显存。因此针对一个 PairFormer Layer 中的两个 Triangle Attention 分别进行重计算。
其次我们对 Triangle Multiplication 进行重计算,因为 Triangle Multiplication 涉及大量 (i,j,k) 维度重排与张量广播,且其激活值规模更大。
最后是 smooth_lddt_loss 计算的重计算(大规模 cdist),smooth_lDDT loss 中一项关键计算为 pairwise distance(cdist),其生成的距离矩阵为 O(L² × d),其中L为原子数量,这与 TriangleAttention 等对应的残基数量不同,原子数量通常比残基数大一个数量级,因此对长序列显存压力极大,我们为 loss 中的该部分单独加入了重计算,使其在反向不需要保留巨大 distance matrix。
实际显存收益
在未开启上述重计算策略时:
• 64GB 显存仅能训练长度 64 的序列。
• 动态显存峰值约为20152 MB。
启用重计算后:
• 显存峰值下降到7025 MB,下降超 60%。
• 最长可支持训练长度提升到 768 tokens。
这一优化是 Protenix MindSpore 版本能够在昇腾A2 平台上成功支持长序列训练的关键技术点之一。
2.2 模型推理优化
在这部分工作中,我们基于对模型性能的分析,逐一找到时间、内存方面的性能瓶颈并予以优化。
Profiling 数据与分析
MindSpore 支持用户使用 Profiler 类对模型的性能进行采集,所获得的 Profiling 数据记录了详细的算子时间线,也包括了算子的显存占用信息。Profiling 数据可以通过 MindInsight 工具进行可视化分析,可以查看详细的算子时间线,以及流之间的调用关系。我们可以精确计算出每个模块的位置及其耗时,并据此来确定这些模块是否需要进一步的优化。例如,下图展示了我们对推理过程中 PairFormer 模块的定位与拆解,为后续的时间、内存的分析提供了框架与引导:
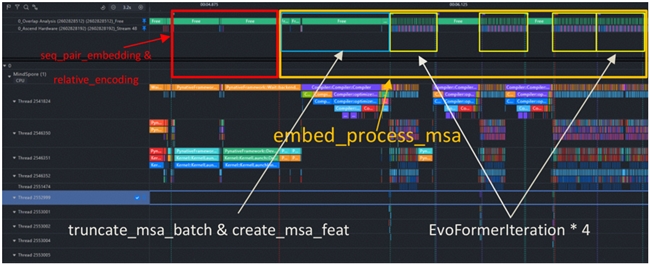
Unfold 算子重构
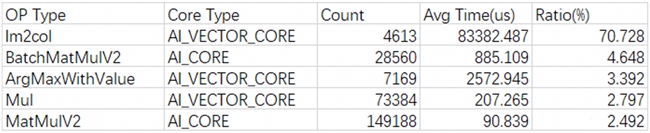
通过模型运行时打印算子运行时长占比,发现 Im2col 占总运行时长最高,高达 70.73%,故需要分析并消减该算子的调用。
定位后可确定为调用 mindspore.ops.unfold 算子引入问题。根据原本 PyTorch 代码逻辑,此处实际使用 torch.Tensor.unfold,其实际与 torch.nn.functional.unfold 行为不同,差异如下:
• Tensor.unfold:返回原始张量的一个视图,该视图包含在指定维度上从张量中提取的所有大小为 size 的切片。
• nn.functional.unfold:把 4-D 图像 (N,C,H,W) 的每个 kernel_size 平面窗拉成一列,输出“二维矩阵”,方便后面用矩阵乘法代替卷积。本质是 im2col 操作,为 im2col 的别名 api。
而 MindSpore 中,Tensor.unfold 与 ms.nn.functional.unfold 实现相同,实际调用为 im2col,因此造成实现差异。故此处整改方案为,使用 MindSpore 实 现 Tensor.unfold 与 torch.Tensor.unfold 相同功能函数进行替换。等价实现后,端到端推理性能提升1倍。后续 MindSpore 实现 Tensor.unfold 算子后可进一步优化显存占用以提升性能。
融合算子的开发与调优
由于 SelfAttention 的显存开销与蛋白质序列长度强相关,且当前对该模块的优化并不完全亲和生物学场景,因此我们选择开发融合算子 EvoformerAttention。对此,我们实施了以下关键改进:
• UB 内存布局重构:消除内存碎片,提升 UB 利用率;
• 消除流同步算子:重构计算流水线,将串行内存拷贝转为并行异步操作;
• 稀疏掩码优化:去除 drop_mask 在 UB 中的显存占用;
• 动态 tiling 调整:基于 UB 剩余容量自适应调整分块大小,显著降低循环开销;以上四个改进总体时间性能提升约 6.5%;
• API 优化:将传统的 Level 1 API 配合显式循环的模式,重构为 Level 0 API 的批量处理接口,单步优化后时间性能提升约 5%。
此外,Protenix 中使用了大量的张量计算,其实现方式均为 Einsum(Einstein Summation,爱因斯坦求和约定),因此该算子对模型整体的性能影响较大。Einsum 中规定的张量缩并运算满足下标表达式
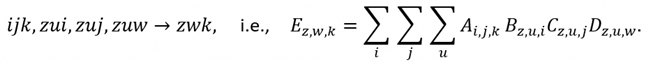
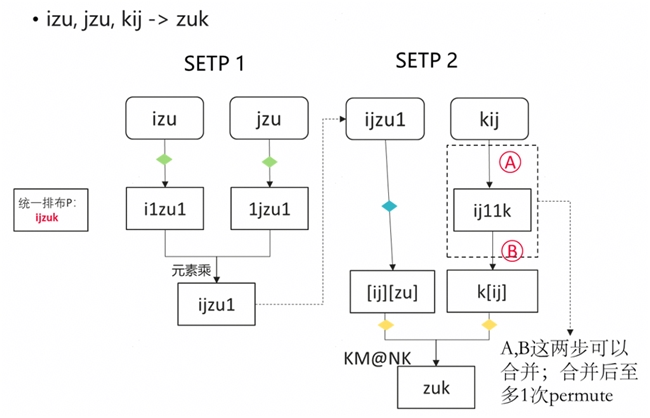
Einsum 高效实现在逻辑上离不开对下标的重排列(permute)。但 permute 操作的时间复杂度是 O(N),我们可以通过优化下标排布,减少或消除显式的 permute 操作,来进一步提升 Einsum 的算子性能。具体操作包括:
• 放弃不必要的 permute 操作,逻辑上改为对下标循环的重排布,并通过 reshape 操作合并下标,以实现批量操作;可将时间复杂度降到O(1);
• 使用 Mindspore 接口:ops.MatMul(transpose_a=False, transpose_b=False),该接口适配了最低两维转置的情况,可以替代符合这种情况下的 permute 操作。
寻找并解决内存瓶颈
经过此前的优化后,Protenix 模型的 MindSpore 实现版本在单张 A2上的推理极限大致为包含 2000 个残基的蛋白质序列,也即推理长度的极限只有 2k。通过分析 2k 长度序列推理的 Profiling 数据、调查模型前期出现的若干个算子,我们发现在模型在 PairFormer 阶段存在大量的内存瓶颈:
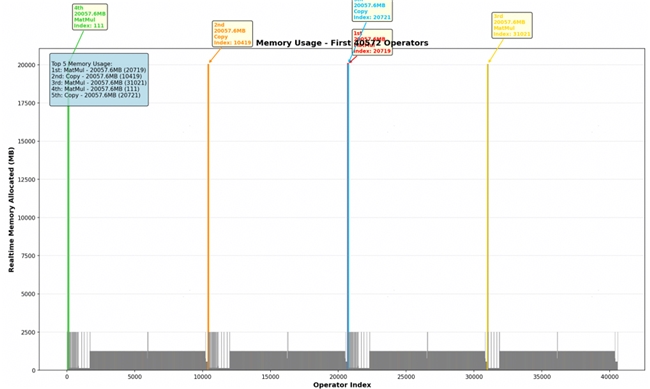
通过对算子的定位我们可以将内存峰值出现的时间与四次 EvoFormer Iteration 相吻合,最终定位出内存瓶颈为该循环中的 outer_product_mean 计算。 该模块主要承担张量的缩并计算(爱因斯坦求和操作) 和一些线性变换,而内存瓶颈正是发生在外积计算当中:
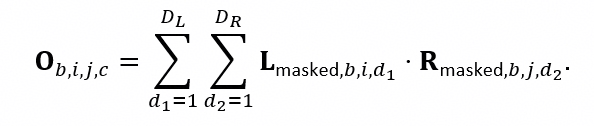
对求和的左侧部分进行分块操作,并调整合适的分块尺寸(chunk_size),成功降低了内存的峰值。我们后续又定位到其他可能导致内存溢出的位置,分别是:
• 位于PairFormer 阶段的 msa_attention,msa_transition 和 triangle_multiplication 计算;
• 位于Diffusion 阶段的 transition_block 计算;
• 位于Confidence 阶段的 ConfidenceHead 和 GridSelfAttention 计算。
关于分块操作对时间、内存以及算法精度上的影响,通过理论推导与实验验证,我们得到以下结论:
• 我们总是避开了 LayerNorm,Softmax 等非线性操作所涉及的维度,因此分块不会影响最终推理的精度;
• 整体而言,分块尺寸与计算时间呈负相关关系,因此可在内存容许的情况下,尽量增大分块尺寸;下图展示了 msa_attention 和 GridSelfAttention 在不同分块下的计算时间;
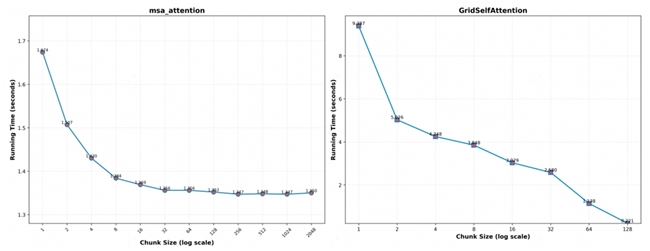
使用以上策略,我们打通了单张 A2 上的 3k 长度序列推理,成功提高了模型的推理极限。
2.3 jit 装饰器与静态图编译
MindSpore 与 PyTorch 的核心差异之一在于:
* PyTorch(Eager Mode)采用运行时逐算子调度,算子粒度小、灵活但存在较高 launch 开销;
* MindSpore 支持通过 **`jit` 装饰器** 将部分模块提前编译为静态图(Graph),在执行时以 **大算子形式一次性下发**,极大减少算子调度成本。
在 Protenix 的 MindSpore 复现中,我们主要对 Transformer 模块进行了 JIT 编译以提升推理与训练效率。这主要是由于 Protenix 的 Transformer 层结构 较为规则,输入维度(hidden size、head_dim、num_heads)均为固定值,适合编译为计算图。在 Diffusion 采样过程中,每步都需要调用 Transformer,共200次,但仅第一次需要编译,后续可以直接复用。以序列长度 109 的蛋白质 5tgy 在 Atlas A2 的端到端推理性能为例(Diffusion 200 steps):
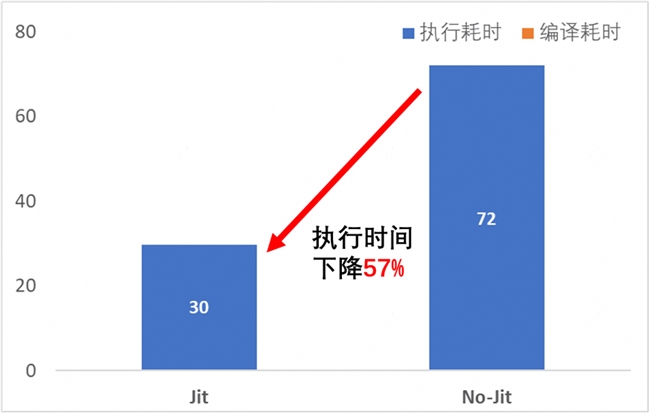
• JIT 编译耗时大约30 s;
• 运行平稳后耗时约41 s;
• 非 JIT 模式下的推理耗时为72 s;
• JIT 模式下端到端加速比达到57%;
总结
我们成功将蛋白质结构预测模型 Protenix 从 PyTorch 迁移至 MindSpore 框架,并在昇腾 A2 平台上实现了高性能训推。针对训练显存瓶颈,我们设计了细粒度的重计算策略,对 Triangle Attention、Triangle Multiplication 等模块进行针对性优化,将动态显存峰值降低 60% 以上,支持 768 长度序列训练。推理优化方面,通过重构 unfold 算子消除冗余 im2col 操作,开发 EvoformerAttention 融合算子,优化 Einsum 实现减少数据移动,并采用分块策略突破outer_product_mean 等模块的内存瓶颈,以及 JIT 编译加速等,将推理长度从 2k 扩展至 3k 以上。我们验证了自主创新计算平台在前沿蛋白质预测任务中的高效性与可行性,为复杂科学计算模型向 MindSpore 生态迁移提供了实践范例。
在蛋白质领域,昇思 AI4S 团队通过算法与自主创新算力的深度协同,使实验室级的前沿AI工具,成为生物医药产业可规模部署的基础设施。昇思 AI4S 团队聚焦于打造面向科学发现的专用 AI 框架,致力于构建科学计算与人工智能融合的新型基础设施。团队支撑范围涵盖了生物信息、地球物理、能源、电磁仿真、计算数学和材料化学等多个领域,未来将进一步打造开源生态并深化基础设施的建造。昇思社区的 AI4S 开源代码仓库可见 https://atomgit.com/mindspore-lab/mindscience.
本次在杭州举办的昇思人工智能框架峰会,将会邀请思想领袖、专家学者、企业领军人物及明星开发者等产学研用代表,共探技术发展趋势、分享创新成果与实践经验。欢迎各界精英共赴前沿之约,携手打造开放、协同、可持续的人工智能框架新生态!













